Mastite bovina: uma pedra no caminho do produtor de leite
O Brasil é o quinto maior produtor mundial de leite, com uma produção de mais de 36 milhões de toneladas por ano (Milkpoint, 2023a). Apesar dessa posição de destaque, um dos fatores que afetam negativamente a produção e a produtividade e, consequentemente, o crescimento da indústria de laticínios no Brasil é a mastite, que é a inflamação da glândula mamária, um problema que gera grandes prejuízos financeiros em todo o mundo.
Para melhor compreensão dos prejuízos gerados pela mastite, Dalanezi et al. (2020) relataram que a perda anual dos produtores de leite nos EUA é de 2 bilhões de dólares, sendo os custos principalmente relacionados à redução na produção e descarte de leite, assim como o descarte e substituição de animais. No Brasil, segundo uma matéria publicada no Milkpoint (2023b), a mastite bovina causa uma perda anual de até 1,75 bilhões de litros de leite.
Como reverter essa situação? Bom, é aí que mora o problema, pois a mastite é uma doença desafiadora, de difícil controle e nem sempre passível de erradicação no rebanho (Arashiro, 2022). Na maioria dos casos, a inflamação acontece devido à invasão do canal do teto por microrganismos patogênicos como bactérias, fungos, algas e vírus, sendo as bactérias responsáveis pela maioria dos casos. Historicamente, as espécies Staphylococcus aureus e Streptococcus agalactiae se mantêm como os principais patógenos da mastite bovina no Brasil, pois estão presentes na maioria dos rebanhos leiteiros do país (Brito et al., 1999; Andretta et al., 2022).
Controlando a mastite bovina nos rebanhos leiteiros
Embora a natureza infecciosa seja predominante, todas as formas de mastite são influenciadas por diversos fatores que podem variar em intensidade dentro do rebanho. Para controlar efetivamente a mastite, é necessário considerar o rebanho como um todo, em vez de apenas cada animal individualmente. Enquanto a mastite clínica ocorre esporadicamente, a mastite subclínica pode estar presente em todos os rebanhos a qualquer momento.
Sendo assim, a fim de reduzir as perdas econômicas proporcionadas pela mastite e promover o bem-estar animal, algumas medidas devem ser adotadas.
Dentre as medidas preventivas para evitar a disseminação de patógenos encontram-se a limpeza adequada dos ambientes, manutenção da higiene na rotina de ordenha, realização da limpeza e higienização dos equipamentos com detergentes específicos, seguindo o tempo de ação e temperatura corretos.
A implementação de um programa de monitoramento regular da saúde da glândula mamária por meio de testes é importante, sendo os testes de caneca de fundo telado em cada ordenha e o California Mastitis Test (CMT), usados para diagnóstico da mastite clínica e subclínica, respectivamente (Resmini et al., 2023).
Mesmo que sejam tomados todos os cuidados para evitar a mastite, ainda é possível a ocorrência da doença, sendo necessário adotar medidas corretivas para controle da infecção, como o descarte do animal, especialmente no caso de mastite crônica, pois os animais nesse caso podem se tornar reservatórios dos agentes causadores da mastite (Ex. S. aureus).
Neste contexto, a antibioticoterapia continua sendo uma das estratégias mais utilizadas para tratamento da mastite clínica nos rebanhos. Entretanto, a presença de resíduos de antibióticos no leite pode significar prejuízos ao produtor devido à necessidade de descarte do leite durante o período de carência. Além disso, o uso de antibióticos eleva o risco da emergência e disseminação de resistência antimicrobiana entre os patógenos, por isso recomenda-se prudência na utilização destas moléculas (OIE, 2008).
Resistência aos antibióticos: um desafio que vai bem além das porteiras
A resistência aos antimicrobianos representa um desafio global para a saúde de forma ampla, ultrapassando a porteira das propriedades leiteiras, pois as bactérias resistentes aos antibióticos, eventualmente presentes nos animais, podem ser transmitidas aos seres humanos através da cadeia alimentar e causar doenças de difícil tratamento ou, até mesmo, se espalharem no ambiente por meio dos resíduos animais.
Dentro dessa complexidade, a ideia de saúde única, promovida pela Organização Mundial da Saúde (OMS), Organização Mundial para Saúde Animal (OIE) e Organização das Nações Unidas para Alimentação e Agricultura (FAO), emerge como um conceito que reconhece a interconexão entre a saúde humana, animal e ambiental. Essa abordagem integrada considera a inseparabilidade desses três domínios da saúde, compreendendo a influência mútua entre eles na preservação da saúde global (CMFV, 2020).
Dados alarmantes foram descritos por Rato e colaboradores (2013), que ao investigarem estreptococos isolados de leite bovino e animais com mastite (n= 108), de diferentes rebanhos do sudoeste de Portugal, constataram a existência de perfis genéticos semelhantes entre estreptococos de origem humana e animal, o que sugeriu uma possível transmissão bovino-humana.
Nas linhagens de S. agalactiae foi detectada resistência à eritromicina, pirlimicina, tetraciclina, gentamicina, estreptomicina e cefoperazona. A análise por PCR revelou que 10 dos 11 isolados resistentes à eritromicina carregavam o gene ermB e apenas um era portador do gene ermA. Nos isolados resistentes à tetraciclina, foram encontrados os genes tetM, tetO e tetK nas proporções de 50%, 45,8% e 100%, respectivamente, como determinantes genéticos da resistência.
Embora o trabalho de Sweileh (2021) não tenha sido com bovinos, também demonstra a importância de monitorarmos a resistência bacteriana a antimicrobianos, pois foi observada a emergência de bactérias resistentes à colistina isoladas de suínos na China. O gene de resistência à colistina 1 (MCR-1) é carreado por um plasmídeo, pequena molécula de DNA com replicação autônoma, que favorece a rápida disseminação da resistência. Essa disseminação é considerada uma ameaça mundial à saúde humana, pois a colistina é um antimicrobiano de última escolha para determinadas enfermidades decorrentes de bactérias multirresistentes.
O perfil de resistência de agentes etiológicos da mastite aos antimicrobianos, bem como os mecanismos moleculares envolvidos na resistência, varia de acordo com a região e entre rebanhos. Ademais, não há um consenso se o nível de resistência entre esses patógenos têm sofrido ou não um aumento acentuado com o passar do tempo e nem como isso pode afetar a saúde humana (Oliver e Murinda, 2012). Acredita-se que as bactérias resistentes multiplicam mais rapidamente mediante o uso de antibióticos devido à pressão de seleção destes que atuam eliminando as bactérias suscetíveis que competem com as resistentes na ausência do quimioterápico.
O uso inadequado ou excessivo de antibióticos permite que as bactérias sobreviventes se reproduzam e transmitam suas características de resistência a bactérias da mesma espécie ou de espécies diferentes, tornando-se mais difíceis de tratar.
No Brasil, pouco se sabe sobre os perfis e os mecanismos moleculares de resistência aos antimicrobianos em patógenos da mastite.
Duarte et al. (2005) investigaram as características de 189 isolados de S. agalactiae resistentes a antimicrobianos de fontes bovinas (n = 38) e humanas (n = 151). As vacas pertenciam a rebanhos distribuídos em dois estados do sudeste do Brasil, Minas Gerais e Rio de Janeiro.
Todas as linhagens eram resistentes à tetraciclina (TET) e 16 (8,5%) eram também resistentes à eritromicina, sendo que 23,7% são isolados bovinos resistentes à TET e 4,6% são isolados humanos resistentes à TET. Os genes tetO, ermB e mreA, foram os mais frequentes entre os isolados bovinos, enquanto os genes tetM e ermA foram os mais prevalentes entre os isolados de humanos.
Considerando a dimensão do Brasil, são necessários estudos com maior número de isolados de S. agalactiae de origem bovina, isoladas de propriedades leiteiras de diferentes estados do país, para melhor representação da realidade do tema em rebanhos brasileiros.
Tendo em vista as informações fornecidas pelos trabalhos citados anteriormente, é necessária e urgente a caracterização e vigilância da resistência por meio da caracterização das estirpes, de modo sistemático e contínuo, em massa e por bacias leiteiras, estados, regiões e país. Desse modo, obteremos informações que permitam avaliar o nível e a evolução da resistência antimicrobiana de patógenos da mastite no Brasil.
Isso poderá auxiliar no controle da resistência e fornecer informações úteis para decisões terapêuticas, bem como melhor compreensão da base molecular da resistência antimicrobiana nesses patógenos (Hernandez et al., 2021).
Monitoramento da resistência: protegendo a eficácia do antibiótico
Monitorar a resistência a antibióticos significa acompanhar e avaliar a capacidade dos microrganismos de resistir aos efeitos dos antibióticos ao longo do tempo. O monitoramento da resistência antimicrobiana faz parte das estratégias para diminuir a emergência de linhagens bacterianas resistentes e otimizar a escolha do antimicrobiano ao realizar o tratamento de alguma doença (Paramasivam et al., 2023; Devadas et al., 2024).
É necessário estabelecer métodos que facilitem a vigilância e o monitoramento da resistência antimicrobiana nos rebanhos do Brasil. A vigilância epidemiológica pode auxiliar na tomada de providências, pois proporciona o conhecimento, a detecção ou prevenção de qualquer mudança nos fatores determinantes e condicionantes de saúde, podendo gerar informações que auxiliam no monitoramento e na definição de protocolos para uso racional de antibióticos em rebanhos, gerando impacto econômico e ambiental.
É sabido que beta-lactâmicos funcionam muito bem para combater S. agalactiae e não há relatos de resistência aos beta-lactâmicos nesses microrganismos, ao contrário de S. aureus. No entanto, com o uso indiscriminado de antibióticos e, consequentemente, a presença desses compostos no ambiente, além da coexistência de S. agalactiae e S. aureus num mesmo rebanho, não se pode descartar a possibilidade de mutação bacteriana, recombinação e transferência horizontal de genes.
Dessa forma, microrganismos antes não resistentes, podem vir a adquirir a resistência. E se houver uma linhagem de S. agalactiae resistente a beta-lactâmicos, quanto tempo levaremos para percebê-la e para tomar providências?
Testes de susceptibilidade antimicrobiana são importantes para verificar a quais antibióticos as bactérias são suscetíveis e identificar possível resistência. As formas mais clássicas de se realizar esses testes são por método de difusão (ex. difusão em discos de antimicrobianos em ágar) e método de diluição (ex. concentração inibitória mínima - MIC). Porém, a difusão em disco continua sendo um dos métodos mais amplamente utilizados, adequado, reprodutível e preciso para fins de monitoramento (Devadas et al., 2024).
Embora o antibiograma por difusão do antimicrobiano seja a forma mais comumente utilizada atualmente para monitoramento da resistência, o uso dessa prática para tratamento da mastite muitas vezes é inviabilizada pelo tempo despendido entre o envio da amostra e a obtenção do resultado, quando na maioria das vezes deseja-se informações rápidas para o início imediato do tratamento com o antibiótico ideal.
Assim, o desenvolvimento de métodos rápidos e confiáveis, preferencialmente que possam ser executados dentro ou em local próximo à propriedade leiteira, é desejável. Esse raciocínio se aplica tanto ao diagnóstico para tratamento como à triagem para monitoramento de linhagens resistentes aos antimicrobianos.
Monitoramento em massa da resistência: o potencial dos marcadores moleculares
Métodos moleculares, que se baseiam na detecção de partes do material genético (DNA ou RNA), têm sido considerados para facilitar o monitoramento da resistência a antimicrobianos. Isso é possível porque a resistência a antibióticos é um fenômeno genético. Existem sequências específicas de DNA, conhecidas como genes, que determinam essa resistência e estão associadas às características observadas.
Deste modo, métodos de detecção de DNA, tais como a Reação em Cadeia da Polimerase (PCR) e suas variantes (RT-PCR, PCR Multiplex) podem ser utilizados para detectar genes relacionados à resistência a antimicrobianos em microrganismos, e assim, permitir ao pesquisador inferir (indiretamente) sobre o fenótipo de resistência no respectivo microrganismo.
Como vantagens em relação ao método convencional (antibiograma), os métodos moleculares permitem resultados mais rápidos, de alto rendimento, com alta sensibilidade e especificidade, maior reprodutibilidade, menor tempo de análise e liberação de resultados, e ainda, não depende de viabilidade do microrganismo.
Além disso, são de simples execução, o custo é acessível e não é imprescindível a extração de DNA (etapa que costuma ser laboriosa e demorada). Contudo, esses métodos apresentam como desvantagens a incapacidade de diferenciar células vivas e mortas, necessidade de conhecimento prévio sobre a base genética e epidemiologia da resistência, e impossibilidade de detectar novos mecanismos de resistência.
Eventualmente a resistência a alguns antibióticos conta com a presença de vários determinantes genéticos alternativos, sendo necessário aumentar o número de marcadores moleculares para melhorar a correspondência entre o resultado genotípico, obtido por métodos moleculares, e o fenótipo do microrganismo.
A aplicação da PCR multiplex na detecção de genes de resistência antimicrobiana tem sido considerada vantajosa, uma vez que permite a amplificação de vários genes em uma única reação com a utilização de múltiplos pares de primers (oligonucleotídeos curtos, composto pelos nucleotídeos adenina, citosina, guanina e timina, que são complementares a uma sequência específica de DNA).
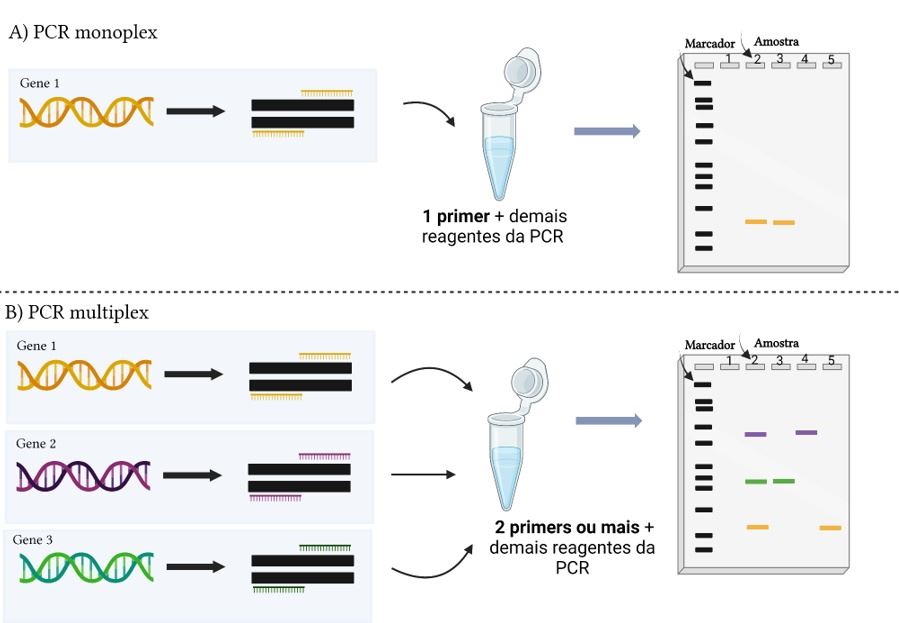
Isso resulta em economia de reagentes e materiais, além de reduzir o tempo de análise, proporcionando uma relação custo-benefício mais favorável (Figura 1) (Albuquerque et al., 2019; Zhu et al., 2019).
Figura 1 – PCR Multiplex. (Fonte: Próprio autor, Criado com BioRender.com).
Legenda: Em A e B, a amostra 1 representa o controle negativo (o controle negativo é uma amostra de DNA do microrganismo que não tem gene de resistência, portanto, não aparecerá bandas), 2 é o controle positivo (o DNA do microrganismo contém todos os genes analisados pelos diferentes pares de primer, portanto aparecerá bandas). Na amostra 3 da parte A, o gene 1 está presente. Na amostra 3 da parte B, o gene 3 está presente. Na amostra 4 da parte B, o gene 2 está presente. Na amostra 5 da parte B, o gene 1 está presente.
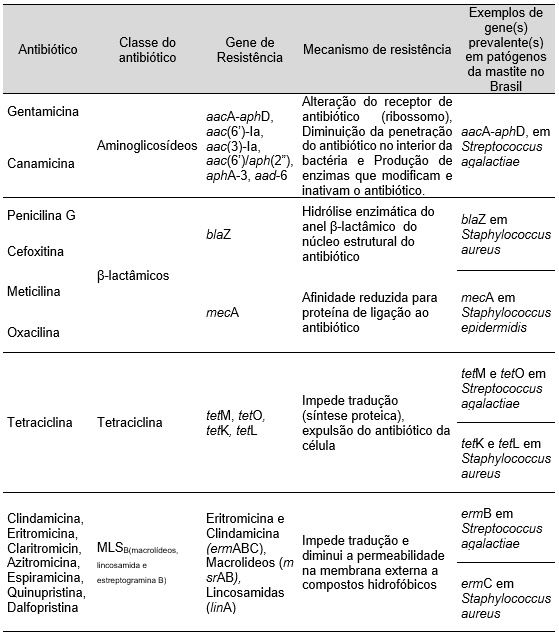
Na tabela 1 podemos ver alguns exemplos dos antibióticos mais utilizados no tratamento da mastite bovina no Brasil, os genes e como ocorre o mecanismo de resistência.
Tabela 1 - Principais classes de antibióticos e mecanismos de resistência em patógenos da mastite (Almeida, 2018).
Gao et al. (2011) desenvolveram uma PCR multiplex para diagnóstico, diretamente em amostras de leite, da mastite causada por Staphylococcus aureus e sua potencial resistência a antibióticos em rebanhos leiteiros da China.
O método de PCR multiplex desenvolvido apresentou alta sensibilidade e especificidade para S. aureus e pôde detectar resistência a antibióticos em linhagens clínicas desse microrganismo e diretamente em amostras de leite mastítico. O método provavelmente pode ser útil para a rápida triagem de resistência a antibióticos em isolados de S. aureus, e deve permitir a administração de um tratamento e/ou controle rápido, mais focado e eficaz, da mastite por essa bactéria.
Entretanto, considerando que as prevalências dos genes de resistência variam de região para região, o método desenvolvido pelos chineses dificilmente poderá ser aplicado em outros países. Portanto, para uso desta estratégia, é crucial o desenvolvimento de métodos de PCR multiplex que permitam detectar genes altamente associados a fenótipos de resistência em patógenos da mastite no Brasil.
Até o momento, apesar da vantagem óbvia do uso da PCR multiplex para a finalidade de monitoramento da resistência, não há métodos baseados na PCR multiplex desenvolvidos e validados no país.
Na maioria dos países que realizam o monitoramento da resistência, o processo envolve as seguintes etapas: definição de amostragem, coleta de amostras de leite nas propriedades, isolamentos e cultivos no laboratório e antibiogramas.
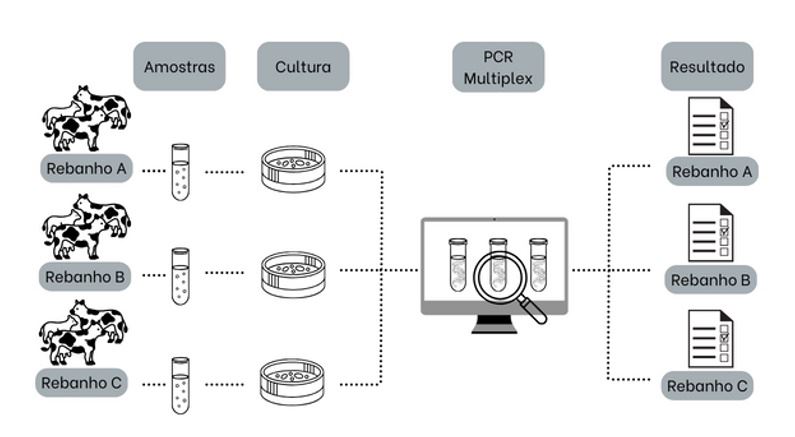
O uso de métodos moleculares pode permitir a análise direta em amostras de leite, eliminando a etapa de cultivo microbiológico, reduzindo o tempo da análise. Entretanto, a complexidade da matriz leite, acrescenta um desafio a mais, na multiplexação da PCR. Considerando a crescente adoção de cultivos na fazenda para diagnóstico da mastite, o método apresenta a possibilidade de usar colônias do microrganismo obtidas diretamente dos cultivos, como fonte do DNA molde na PCR, e isto também requer padronização e validação (Figura 2).
Deste modo, o monitoramento pode se valer do cultivo na fazenda para acelerar a obtenção dos perfis genéticos de resistência a antimicrobianos dos microrganismos, bem como aproveitar a identificação taxonômica local. A detecção dos genes de resistência, poderia ser feita em laboratórios convencionais equipados para realização de PCR e eletroforese, ou alternativamente, poder-se-ia montar uma unidade móvel suficientemente equipada para visitas às fazendas e realização de todas as etapas da obtenção de genótipos bacterianos.
Figura 2 - Proposta da PCR Multiplex para o monitoramento de genes de resistência a antimicrobianos diretamente na fazenda.
Fonte: Próprio autor
Por que os marcadores moleculares ainda não podem ser utilizados em massa para o monitoramento de genes de resistência no contexto da mastite bovina?
Um requisito essencial para a utilização de marcadores moleculares para monitorar resistência é o conhecimento de sua distribuição entre isolados, para seleção daqueles com elevada associação do marcador ao fenótipo de resistência. Desse modo, nem todo marcador de resistência é suficientemente informativo para esta análise.
Um marcador ideal é aquele que apresenta 100% de associação ao fenótipo de resistência em uma população bacteriana. Muitas vezes isso não é possível porque algumas resistências podem apresentar mais de um mecanismo, como por exemplo, a resistência à tetraciclina que é mediada por genes tet em patógenos da mastite (principalmente tetM, tetN, tetO, tetK, tetL). Assim, estudos são necessários para identificar marcadores mais prevalentes, o que pode variar de acordo com a espécie, região e ao longo do tempo.
Eventualmente, há situações em que o dado referente ao fenótipo de resistência não corresponde ao dado genotípico. Ou seja, o fato de detectar o gene de resistência ao antibiótico não garante que a bactéria seja resistente ao antimicrobiano.
Assim como, a ausência do gene nem sempre garante a susceptibilidade ao antibiótico, pois existe a possibilidade de ocorrência de mecanismos substitutos codificados por genes alternativos. Entretanto, o estudo de Ivanovic et al. (2023) demonstrou que há elevada correspondência entre o fenótipo de resistência a beta lactâmicos e a presença do gene blaZ em Staphylococcus aureus isolados de rebanhos de países europeus. Foi relatado que a especificidade dos testes fenotípicos e genotípicos foi de 100%, com todas as linhagens suscetíveis à penicilina (pelo método de disco de difusão e MIC) desprovidas do gene blaZ e a sensibilidade (fração de linhagens resistentes à penicilina/positivas para blaZ) foi de 68%. Estudos como este são ainda escassos para a maior parte dos patógenos da mastite bovina, principalmente em relação às resistências mais comuns encontradas tanto no Brasil quanto globalmente.
Hernandez et al. (2021) avaliaram a susceptibilidade antimicrobiana e a presença de genes de resistência relacionados aos antimicrobianos em S. agalactiae que circulam entre bovinos com mastite em fazendas da Argentina.
Foram utilizados 8 antimicrobianos empregados na medicina animal e humana, a fim de obterem informações importantes no contexto de saúde única. Ao utilizarem tanto a abordagem fenotípica quanto genotípica, foi constatada uma correlação média de 65,62% entre fenótipos de resistência a tetraciclina, eritromicina, canamicina e pirlimicina/clindamicina e a presença dos genes correspondentes tetM e tetO relacionados a tetraciclina, ermB a eritromicina, aphA3 a canamicina e lnuB a pirlimicina/clindamicina.
Sendo que, a correlação entre o fenótipo e genótipo de tetraciclina e eritromicina foi de 100%, enquanto para canamicina e pirlimicina/clindamicina foi de 37,5% e 25%, respectivamente. Em contrapartida, 50% das linhagens que mostraram susceptibilidade fenotípica para todas as classes de antimicrobianos carregavam alguns genes de resistência.
Dados como esses demonstram a importância de monitorar a propagação de estirpes utilizando as duas diferentes abordagens, e que estudos podem ser realizados a fim de obter correlações melhores (>60%) entre fenótipo e genótipo.
Embora os marcadores moleculares tenham suas limitações em relação à determinação do fenótipo de resistência bacteriana e à viabilidade dos patógenos, métodos confiáveis e validados com base nesses marcadores podem fornecer informações valiosas sobre a resistência a antimicrobianos. Essas informações são essenciais devido à coexistência de diferentes microrganismos (patogênicos e não patogênicos) em uma fazenda leiteira e à possibilidade de transferência de sequências de DNA entre eles por meio de mecanismos genéticos.
Além disso, o acúmulo de dados relacionados a esse tema por região pode viabilizar estudos de análise de risco e até na definição de protocolos de uso racional de antibióticos, contribuindo para estudos de saúde única.
Assim, a “simples” presença do gene de resistência pode ser considerada um risco em potencial, especialmente, se o mesmo for detectado no rebanho, visto que esse gene pode ser incorporado por um patógeno e conferir-lhe o fenótipo de resistência. Além disso, a falta de um marcador confiável em um isolado bacteriano, pode ser importante para decidir o uso de um antimicrobiano, pois um marcador altamente específico pode indicar a susceptibilidade da linhagem bacteriana (Ivanovic et al., 2023).
Além disso, a PCR permite a detecção em massa de genes de resistência de modo rápido e a baixo custo, diferentemente dos métodos convencionais. E o recomendável é que sejam utilizados métodos baseados em marcadores cuja associação com fenótipo de resistência seja satisfatória, o que necessita ser definido por estudos prévios. Ademais é necessário que tais métodos sejam avaliados e validados em diferentes populações e regiões bem como em diferentes tempos.
Os perfis de resistência obtidos pela PCR poderão ser utilizados em associação com métodos que permitem a discriminação sub específica de isolados bacterianos para monitorar de perto a propagação de estirpes resistentes e multirresistentes e explorar os mecanismos moleculares responsáveis pela resistência antimicrobiana.
Diante desse cenário, é necessário focar em pesquisas que visam padronizar e validar métodos moleculares, especialmente baseados em PCR, para monitoramento da resistência a antimicrobianos, a fim de obtermos métodos robustos e confiáveis para monitorar os marcadores moleculares nos rebanhos bovinos leiteiros.
Afinal de contas, é confiável ou não monitorar a resistência por marcadores moleculares?
Possivelmente no futuro, sim. Reconhecemos que a melhor forma para obtenção dos dados brutos ainda é a avaliação fenotípica dos patógenos quanto à resistência aos antibióticos. Entretanto, o acúmulo de conhecimento epidemiológico da resistência antimicrobiana por meio de estudos espacialmente e temporalmente bem delineados (municípios, bacias leiteiras, estados, regiões, etc.) permite a perspectiva de desenvolver métodos bem padronizados e customizados para cada região, visto que, um método único, dificilmente será eficaz para tal empreitada num país continental como o Brasil.
Para isso, é necessário uma rede robusta de laboratórios minimamente equipados para a vigilância e monitoramento da resistência aos antimicrobianos. Além disso, é essencial um sistema simples e eficiente para o compartilhamento e armazenamento de dados referentes aos perfis moleculares de resistência aos antimicrobianos, bem como, a garantia de proteção dos dados de cada produtor. Desse modo, possibilitar-se-á a elaboração de relatórios periódicos com acesso público.
Autores
Paula Aparecida Azevedo Almeida
Bianca de Oliveira Hosken
Joice Fátima Moreira Silva
Mariluce Jaguraba de Jesus Almeida
Márcio Roberto Silva
Guilherme Nunes de Souza
Geraldo Márcio da Costa
João Batista Ribeiro
Referências
Arashiro, E.K.N.; Teodoro, V.A.M.; Miguel, E.M. Mastite bovina: importância econômica e tecnológica. Ciência do leite. Disponível em <https://cienciadoleite.com.br/noticia/5221/mastite-bovina-importancia-economica-e-tecnologica>. Acesso em 23 de outubro de 2023. 2022.
Albuquerque, R.C. et al. Multiplex-PCR for diagnosis of bacterial meningitis. Brazilian Journal of Microbiology, v.50, p. 435–443. 2019. https://doi.org/10.1007/s42770-019-00055-9
Almeida, P.A.A. Desenvolvimento e avaliação de método molecular baseado em PCR multiplex para detecção de genes de resistência a antimicrobianos relevantes para o controle da mastite bovina. 2018. 82f. Dissertação (Mestrado Profissional em Ciência e Tecnologia do Leite e Derivados) - Faculdade de Farmácia, Universidade Federal de Juiz de Fora - UFJF, Juiz de Fora, Disponível em <https://repositorio.ufjf.br/jspui/browse?type=author&value=Almeida%2C+Paula+Aparecida+Azevedo>. Acesso em 23 de outubro de 2023. 2018.
Andretta, M.; Call, D.R.; Nero, L.A. Insights into antibiotic use in Brazilian dairy production. International Journal of Dairy Technology, v.76, n.1. 2022. https://doi.org/10.1111/1471-0307.12914
Brito, M.A.V.P.; Brito, J.R.F.; Ribeiro, M.T., Veiga, V.M.O. Padrão de infecção intramamária em rebanhos leiteiros: exame de todos os quartos mamários das vacas em lactação. Arq.Bras. Med. Vet. Zootec.,v. 51, n. 2, 129-135, 1999.
CFMV. Conselho Federal de Medicina Veterinária. Conceito: Saúde Única. Disponível em <https://www.cfmv.gov.br/wp-content/uploads/2020/01/folder-saude-unica.pdf>. Acesso em 22 de novembro de 2023. 2020.
Dalanezi, F.M. et al. Influence of pathogens causing clinical mastitis on reproductive variables of dairy cows. Journal of Dairy Science, v. 103, n. 4, p. 3648-3655. 2020. doi: 10.3168/jds.2019-16841. 2020.
Devadas S, Zakaria Z, Shariff M, Bhassu S, Karim M, Natrah I. Methodologies and standards for monitoring antimicrobial use and antimicrobial resistance in shrimp aquaculture, Aquaculture, v. 579. <https://doi.org/10.1016/j.aquaculture.2023.740216>. 2024.
Duarte, R.S. et al. Distribuição de genes relacionados à resistência antimicrobiana e à virulência entre estreptococos brasileiros do grupo B recuperados de fontes bovinas e humanas. Antimicrobiano. Agents Quimother. v.49, p.97–103. 2005 doi: 10.1128/AAC.49.1.97-103.2005
Gao, J. et al. Development of multiplex polymerase chain reaction assay for rapid detection of Staphylococcus aureus and selected antibiotic resistance genes in bovine mastitic milk samples. Journal of Veterinary Diagnostic Investigation, v. 23, p. 894-901, 2011. doi: 10.1177/1040638711416964
Hernandez, L. et al. Multidrug resistance and molecular characterization of Streptococcus agalactiae isolates from dairy cattle with mastitis. Front Cell Infect Microbiol. v.11 2021. doi: 10.3389/fcimb.2021.647324.
Ivanovic, I., Boss, R., Romanò, A., Guédon, E., Le-Loir, Y., Luini, M., Graber, H.U. Penicillin resistance in bovine Staphylococcus aureus: Genomic evaluation of the discrepancy between phenotypic and molecular test methods. Journal of Dairy Science, v.106, n.1, p. 462-475. 2022. https://doi.org/10.3168/jds.2022-22158.
Milkpoint. Relatório destaca números da produção de leite mundial. Disponivel em <https://www.milkpoint.com.br/noticias-e-mercado/giro-noticias/quem-e-o-maior-produtor-mundial-o-top5-o-maior-exportador-descubra-aqui-233550/>. Acesso em 22 de outubro de 2023. 2023a.
Milkpoint. Mastite bovina causa prejuízo anual de até 1,75 bilhão de litros de leite no Brasil. Disponivel em: <https://www.milkpoint.com.br/empresas/novidades-parceiros/mastite-bovina-causa-prejuizo-anual-de-ate-175-bilhao-de-litros-de-leite-no-brasil-235063/> . Acesso em: 23 de outubro de 2023. 2023b.
OIE. World Organization for Animal Health. Risk assessment for antimicrobial resistance arising from the use of antimicrobials in animals. In: OIE. Terrestrial animal health code. Paris: OIE, 2008. (Capítulo 6.8. p. 16). Disponível em <httD://www.oie.intlena/normes/Mcode/ensommaire.htm.> Acesso em 13 de Novembro de 2023.
Oliver, S. P., Murinda, S. E. Antimicrobial Resistance of Mastitis Pathogens. Vet. Clin. Food Anim. v. 28, p.165185, 2012.
Paramasivam, R. et al. Is AMR in Dairy Products a Threat to Human Health? An Updated Review on the Origin, Prevention, Treatment, and Economic Impacts of Subclinical Mastitis. Infection and Drug Resistance, v. 6, n. 16, pp.155-178. 2023. doi: 10.2147/IDR.S384776.
Rato, M. G. et al. Antimicrobial resistance and molecular epidemiology of streptococci from bovine mastitis. Veterinary Microbiology. v.161, p.3-4, 2013. doi: 10.1016/j.vetmic.2012.07.043.
Resmini, G. B et al. Prevenção e controle da mastite bovina. Anais da Feira de Ciências, Tecnologia, Arte e Cultura do Instituto Federal Catarinense do Campus Concórdia /Informativos, v. 6. 2023. Disponível em: <https://publicacoes.ifc.edu.br/index.php/fecitac/article/view/4393/3621>. Acesso em: 09 de novembro de 2023.
Sweileh, W. Global research publications on irrational use of antimicrobials: call for more research to contain antimicrobial resistance. Global Health, v. 17, p. 94. 2021. doi: 10.1186/s12992-021-00754-9
Zhu, G.Q., Li, L., Ohiolei, J.A. et al. A multiplex PCR assay for the simultaneous detection of Taenia hydatigena, T. multiceps, T. pisiformis, and Dipylidium caninum infections. BMC Infectious Diseases, v.19, p.854. 2019. doi: 10.1186/s12879-019-4512-3.

 FAÇA SEU LOGIN E ACESSE CONTEÚDOS EXCLUSIVOS
FAÇA SEU LOGIN E ACESSE CONTEÚDOS EXCLUSIVOS